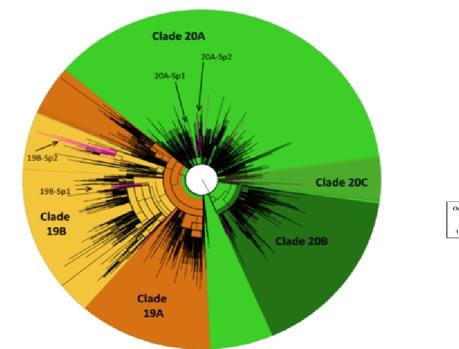
El inicio de la epidemia por coronavirus SARS-CoV-2 en España estuvo marcado por múltiples introducciones de diferentes variantes del virus, por la dispersión durante el mes de febrero (o incluso antes) de al menos cuatro de esas variantes de forma silenciosa por la completa sustitución de variantes que carecían de la mutación D614G durante las siguientes semanas, probablemente debido a la ventaja que supone dicha mutación al ser más infectiva. Lo han visto investigadores del ISCIII en un estudio que ha analizado la evolución y diversidad genética del coronavirus SARS-CoV-2 en España al inicio de la pandemia, cuyos resultados, que se publican en la revista ‘Journal of Virology’, permiten establecer un ‘mapa’ de la transmisión geográfica de las diferentes variantes genéticas del virus, una información que ayuda a explicar la dispersión del SARS-CoV-2 y su evolución a lo largo de la pandemia. «A finales de febrero ya pudieron detectarse representantes de los principales grupos del coronavirus», señala a ABC Salud Francisco Díez, uno de los autores del artículo. En concreto, «detectamos variantes del grupo 19B en Valencia, Andalucía, País Vasco y La Rioja; del 20A en Madrid, Castilla y León, Canarias y Extremadura; del 20B en Valencia, Canarias y Baleares; y del 19A en Andalucía». Además, otro de los principales resultados del trabajo es que al menos 4 de las 62 introducciones identificadas en nuestro país al principio de la epidemia, ya se estaban expandiendo de forma silenciosa a mediados de febrero y probablemente unas pocas semanas antes. «Estas 4 variantes -explica-, que no estaban relacionadas con los dos primeros casos de infección por coronavirus detectadas en España en La Gomera y en Mallorca a principios de febrero, se expandieron a su vez a diferentes países de todo el mundo». Estas variantes comenzaron a expandirse debido a un ‘efecto fundador’; es decir, «las variantes que primero llegaban eran las que se acababan imponiendo». Estas variantes comenzaron a expandirse debido a un ‘efecto fundador’; es decir, «las variantes que primero llegaban eran las que se acababan imponiendo». Sin embargo, a medida que fueron pasando las semanas las variantes sin la mutación D614G pasaron a ser minoritarias y posteriormente desaparecieron. En ensayos in vitro los investigadores han podido asociar la mutación D614G con una mayor capacidad infectiva, hecho que podría explicar la desaparición de las variantes que carecen de dicha mutación. Análisis filogenético de la evolución del virusLa situación observada en España, añade este investigador, es diferente a lo vista en otros países europeos donde la frecuencia de estas variantes durante los primeros meses de la epidemia fue mucho menor. Sin embargo, la situación cambió rápidamente y la frecuencia de estas variantes ha ido disminuyendo a lo largo de la epidemia y ha ido sustituyéndose por variantes que sí presentaban la mutación D614G, hasta el punto de que prácticamente todo lo que se secuencia ahora mismo tiene dicha mutación. «Monitorizar la diversidad genética presente en España para poder entender las dinámicas de transmisión del virus y mejorar nuestro conocimiento sobre el origen y dispersión del coronavirus a nivel local, nacional e internacional» Para Díez, este tipo de trabajos evidencian la necesidad de «monitorizar la diversidad genética presente en España para poder entender las dinámicas de transmisión del virus y mejorar nuestro conocimiento sobre el origen y dispersión del coronavirus a nivel local, nacional e internacional». Y añade: «toda esta información resulta fundamental para evaluar el efecto de medidas de intervención encaminadas a frenar la dispersión del virus como los confinamientos o las cuarentenas».
[Solo los usuarios registrados y activados pueden ver los enlaces. Haga clic aquí para registrarse ... ]


 El inicio de la epidemia por coronavirus SARS-CoV-2 en España estuvo marcado por múltiples introducciones de diferentes variantes del virus, por la dispersión durante el mes de febrero (o incluso antes) de al menos cuatro de esas variantes de forma silenciosa por la completa sustitución de variantes que carecían de la mutación D614G durante las siguientes semanas, probablemente debido a la ventaja que supone dicha mutación al ser más infectiva. Lo han visto investigadores del ISCIII en un estudio que ha analizado la evolución y diversidad genética del coronavirus SARS-CoV-2 en España al inicio de la pandemia, cuyos resultados, que se publican en la revista ‘Journal of Virology’, permiten establecer un ‘mapa’ de la transmisión geográfica de las diferentes variantes genéticas del virus, una información que ayuda a explicar la dispersión del SARS-CoV-2 y su evolución a lo largo de la pandemia. «A finales de febrero ya pudieron detectarse representantes de los principales grupos del coronavirus», señala a ABC Salud Francisco Díez, uno de los autores del artículo. En concreto, «detectamos variantes del grupo 19B en Valencia, Andalucía, País Vasco y La Rioja; del 20A en Madrid, Castilla y León, Canarias y Extremadura; del 20B en Valencia, Canarias y Baleares; y del 19A en Andalucía». Además, otro de los principales resultados del trabajo es que al menos 4 de las 62 introducciones identificadas en nuestro país al principio de la epidemia, ya se estaban expandiendo de forma silenciosa a mediados de febrero y probablemente unas pocas semanas antes. «Estas 4 variantes -explica-, que no estaban relacionadas con los dos primeros casos de infección por coronavirus detectadas en España en La Gomera y en Mallorca a principios de febrero, se expandieron a su vez a diferentes países de todo el mundo». Estas variantes comenzaron a expandirse debido a un ‘efecto fundador’; es decir, «las variantes que primero llegaban eran las que se acababan imponiendo». Estas variantes comenzaron a expandirse debido a un ‘efecto fundador’; es decir, «las variantes que primero llegaban eran las que se acababan imponiendo». Sin embargo, a medida que fueron pasando las semanas las variantes sin la mutación D614G pasaron a ser minoritarias y posteriormente desaparecieron. En ensayos in vitro los investigadores han podido asociar la mutación D614G con una mayor capacidad infectiva, hecho que podría explicar la desaparición de las variantes que carecen de dicha mutación. Análisis filogenético de la evolución del virusLa situación observada en España, añade este investigador, es diferente a lo vista en otros países europeos donde la frecuencia de estas variantes durante los primeros meses de la epidemia fue mucho menor. Sin embargo, la situación cambió rápidamente y la frecuencia de estas variantes ha ido disminuyendo a lo largo de la epidemia y ha ido sustituyéndose por variantes que sí presentaban la mutación D614G, hasta el punto de que prácticamente todo lo que se secuencia ahora mismo tiene dicha mutación. «Monitorizar la diversidad genética presente en España para poder entender las dinámicas de transmisión del virus y mejorar nuestro conocimiento sobre el origen y dispersión del coronavirus a nivel local, nacional e internacional» Para Díez, este tipo de trabajos evidencian la necesidad de «monitorizar la diversidad genética presente en España para poder entender las dinámicas de transmisión del virus y mejorar nuestro conocimiento sobre el origen y dispersión del coronavirus a nivel local, nacional e internacional». Y añade: «toda esta información resulta fundamental para evaluar el efecto de medidas de intervención encaminadas a frenar la dispersión del virus como los confinamientos o las cuarentenas».
El inicio de la epidemia por coronavirus SARS-CoV-2 en España estuvo marcado por múltiples introducciones de diferentes variantes del virus, por la dispersión durante el mes de febrero (o incluso antes) de al menos cuatro de esas variantes de forma silenciosa por la completa sustitución de variantes que carecían de la mutación D614G durante las siguientes semanas, probablemente debido a la ventaja que supone dicha mutación al ser más infectiva. Lo han visto investigadores del ISCIII en un estudio que ha analizado la evolución y diversidad genética del coronavirus SARS-CoV-2 en España al inicio de la pandemia, cuyos resultados, que se publican en la revista ‘Journal of Virology’, permiten establecer un ‘mapa’ de la transmisión geográfica de las diferentes variantes genéticas del virus, una información que ayuda a explicar la dispersión del SARS-CoV-2 y su evolución a lo largo de la pandemia. «A finales de febrero ya pudieron detectarse representantes de los principales grupos del coronavirus», señala a ABC Salud Francisco Díez, uno de los autores del artículo. En concreto, «detectamos variantes del grupo 19B en Valencia, Andalucía, País Vasco y La Rioja; del 20A en Madrid, Castilla y León, Canarias y Extremadura; del 20B en Valencia, Canarias y Baleares; y del 19A en Andalucía». Además, otro de los principales resultados del trabajo es que al menos 4 de las 62 introducciones identificadas en nuestro país al principio de la epidemia, ya se estaban expandiendo de forma silenciosa a mediados de febrero y probablemente unas pocas semanas antes. «Estas 4 variantes -explica-, que no estaban relacionadas con los dos primeros casos de infección por coronavirus detectadas en España en La Gomera y en Mallorca a principios de febrero, se expandieron a su vez a diferentes países de todo el mundo». Estas variantes comenzaron a expandirse debido a un ‘efecto fundador’; es decir, «las variantes que primero llegaban eran las que se acababan imponiendo». Estas variantes comenzaron a expandirse debido a un ‘efecto fundador’; es decir, «las variantes que primero llegaban eran las que se acababan imponiendo». Sin embargo, a medida que fueron pasando las semanas las variantes sin la mutación D614G pasaron a ser minoritarias y posteriormente desaparecieron. En ensayos in vitro los investigadores han podido asociar la mutación D614G con una mayor capacidad infectiva, hecho que podría explicar la desaparición de las variantes que carecen de dicha mutación. Análisis filogenético de la evolución del virusLa situación observada en España, añade este investigador, es diferente a lo vista en otros países europeos donde la frecuencia de estas variantes durante los primeros meses de la epidemia fue mucho menor. Sin embargo, la situación cambió rápidamente y la frecuencia de estas variantes ha ido disminuyendo a lo largo de la epidemia y ha ido sustituyéndose por variantes que sí presentaban la mutación D614G, hasta el punto de que prácticamente todo lo que se secuencia ahora mismo tiene dicha mutación. «Monitorizar la diversidad genética presente en España para poder entender las dinámicas de transmisión del virus y mejorar nuestro conocimiento sobre el origen y dispersión del coronavirus a nivel local, nacional e internacional» Para Díez, este tipo de trabajos evidencian la necesidad de «monitorizar la diversidad genética presente en España para poder entender las dinámicas de transmisión del virus y mejorar nuestro conocimiento sobre el origen y dispersión del coronavirus a nivel local, nacional e internacional». Y añade: «toda esta información resulta fundamental para evaluar el efecto de medidas de intervención encaminadas a frenar la dispersión del virus como los confinamientos o las cuarentenas».

